sklearn 是 Python 中最核心的机器学习库之一,它提供了简单、高效的工具用于数据挖掘和数据分析,其聚类模块 sklearn.cluster 包含了多种经典的聚类算法。
目录
- 聚类是什么?
- 聚类算法的选择
- K-Means 聚类
- 原理
- 代码实现
- 如何确定 K 值?
- DBSCAN 聚类
- 原理
- 代码实现
- 优点与缺点
- 层次聚类
- 原理
- 代码实现
- 高斯混合模型
- 原理
- 代码实现
- 聚类评估指标
- 内部指标 (轮廓系数 Calinski-Harabasz)
- 外部指标 (兰德指数 ARI, 互信息调整 MI)
- 聚类工作流总结
聚类是什么?
聚类是一种无监督学习方法,其目标是将数据集中的样本划分为若干个不同的组(称为“簇”或“Cluster”),使得:
- 同一簇内的样本彼此相似。
- 不同簇间的样本彼此相异。
与分类任务不同,聚类算法在训练时没有预先定义的标签,它完全是根据数据本身的内在结构或分布来进行划分的。
聚类算法的选择
没有一种“最好”的聚类算法,选择哪种算法取决于你的数据特性和分析目标。
| 算法 | 原理 | 优点 | 缺点 | 适用场景 |
|---|---|---|---|---|
| K-Means | 基于距离,将数据分配到最近的“质心” | 简单、快速、可解释性强 | 需要预先指定 K 值;对初始质心敏感;对非凸形状簇效果差 | 凸形簇、大小相似的簇 |
| DBSCAN | 基于密度,将高密度区域划分为簇 | 无需指定 K 值;能发现任意形状的簇;可识别噪声点 | 对参数 eps 和 min_samples 敏感;对密度差异大的簇效果差 |
有噪声、任意形状的簇 |
| 层次聚类 | 通过计算样本间的距离,逐步合并或分裂簇 | 不需要预先指定 K 值;可生成树状图,直观展示簇的层次关系 | 计算复杂度高 (O(n²)),不适合大数据集 | 需要了解数据层次结构的场景 |
| 高斯混合模型 | 假设数据由多个高斯分布混合生成 | 软聚类(样本可属于多个簇,有概率);提供了丰富的概率框架 | 计算相对复杂;对初始值敏感 | 数据分布符合高斯混合模型、需要概率输出的场景 |
K-Means 聚类
原理
K-Means 是最简单、最流行的聚类算法之一。
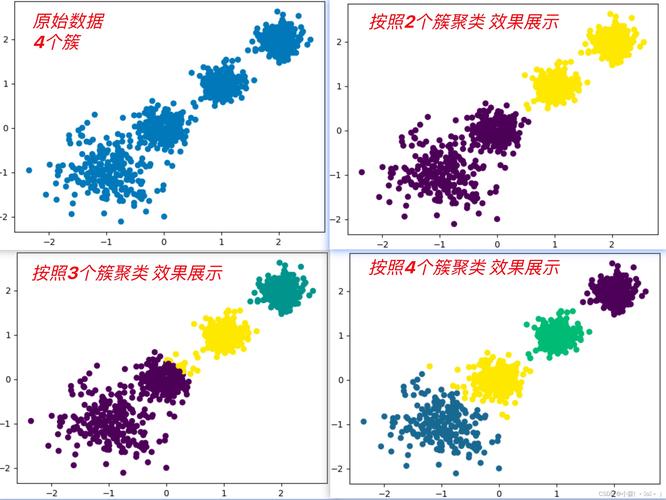
- 初始化: 随机选择 K 个数据点作为初始的“质心”(Centroid)。
- 分配: 计算每个数据点到所有 K 个质心的距离,并将每个数据点分配给距离最近的质心所在的簇。
- 更新: 重新计算每个簇中所有数据点的均值,并将该均值作为新的质心。
- 重复: 重复步骤 2 和 3,直到质心的位置不再发生显著变化(或达到最大迭代次数)。
代码实现
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs
# 1. 生成示例数据
# make_blobs可以生成用于聚类的各向同性高斯 blobs
X, y_true = make_blobs(n_samples=300, centers=4, cluster_std=0.70, random_state=0)
# 2. 创建并训练 K-Means 模型
# n_clusters: 簇的数量
# n_init: 使用不同的质心初始化运行算法的次数,选择最佳结果
# random_state: 随机种子,保证结果可复现
kmeans = KMeans(n_clusters=4, n_init=10, random_state=0)
kmeans.fit(X)
# 3. 获取聚类结果
# labels_: 每个样本所属的簇的标签
# cluster_centers_: 每个簇的质心坐标
labels = kmeans.labels_
centers = kmeans.cluster_centers_
# 4. 可视化结果
plt.figure(figsize=(10, 6))
# 绘制数据点,用不同颜色表示不同簇
plt.scatter(X[:, 0], X[:, 1], c=labels, s=50, cmap='viridis', alpha=0.7)
# 绘制质心
plt.scatter(centers[:, 0], centers[:, 1], c='red', s=200, marker='X', label='Centroids')
'K-Means Clustering Result')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.legend()
plt.grid(True)
plt.show()
如何确定 K 值?
确定最佳的簇数量 K 是一个关键问题,常用的方法是肘部法则。
- 计算不同 K 值下,每个样本到其所属簇质心的距离之和(称为簇内平方和,Inertia 或 WCSS)。
- 绘制 K 值与 Inertia 的关系图。
- 图形通常会呈现一个“肘部”形状,当 Inertia 下降趋势开始变缓时,对应的 K 值就是一个较好的选择。
inertias = []
K_range = range(1, 11) # 测试 K 从 1 到 10
for k in K_range:
kmeans = KMeans(n_clusters=k, n_init=10, random_state=0)
kmeans.fit(X)
inertias.append(kmeans.inertia_)
# 绘制肘部法则图
plt.figure(figsize=(10, 6))
plt.plot(K_range, inertias, 'bo-')
plt.xlabel('Number of clusters (K)')
plt.ylabel('Inertia')'Elbow Method for Optimal K')
plt.grid(True)
plt.show()
在这个图中,你可以看到在 K=4 时,曲线的“肘部”非常明显,因此选择 K=4 是合理的。
DBSCAN 聚类
原理
DBSCAN (Density-Based Spatial Clustering of Applications with Noise) 是一种基于密度的算法。
- 核心点: 在其
eps邻域内至少有min_samples个点的点。 - 边界点: 不是核心点,但在某个核心点的邻域内。
- 噪声点: 既不是核心点也不是边界点的点。
算法通过连接核心点来形成簇,并将噪声点单独分出来。
代码实现
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import DBSCAN
from sklearn.datasets import make_moons
# 1. 生成非凸形状的示例数据
X, y_true = make_moons(n_samples=300, noise=0.05, random_state=0)
# 2. 创建并训练 DBSCAN 模型
# eps: 一个点的邻域半径
# min_samples: 成为核心点所需的最小邻域点数
dbscan = DBSCAN(eps=0.2, min_samples=5)
dbscan.fit(X)
# 3. 获取聚类结果
# labels_: -1 代表噪声点
labels = dbscan.labels_
n_clusters = len(set(labels)) - (1 if -1 in labels else 0) # 计算簇的数量
n_noise = list(labels).count(-1) # 计算噪声点的数量
print(f"Estimated number of clusters: {n_clusters}")
print(f"Estimated number of noise points: {n_noise}")
# 4. 可视化结果
plt.figure(figsize=(10, 6))
# 为每个簇分配一个颜色,噪声点用黑色
unique_labels = set(labels)
colors = [plt.cm.Spectral(each) for each in np.linspace(0, 1, len(unique_labels))]
for k, col in zip(unique_labels, colors):
if k == -1:
# 黑色用于噪声点
col = [0, 0, 0, 1]
class_member_mask = (labels == k)
xy = X[class_member_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
markeredgecolor='k', markersize=6 if k != -1 else 10)
'DBSCAN Clustering')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.grid(True)
plt.show()
层次聚类
原理
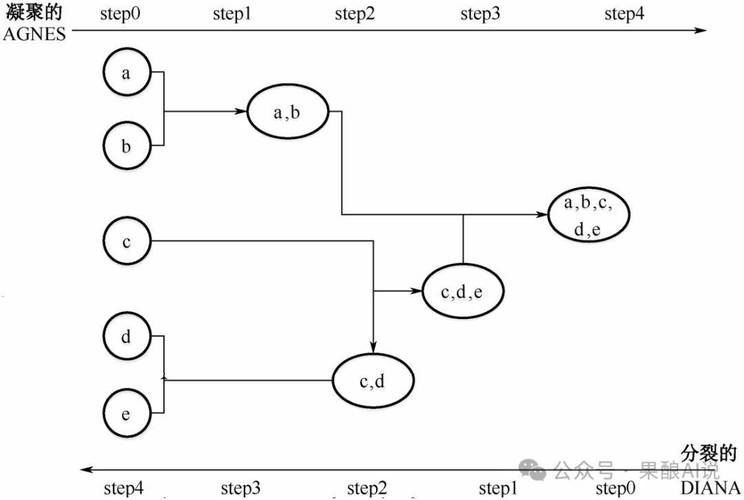
层次聚类有两种主要方法:
- 凝聚式: 从每个点作为一个簇开始,逐步合并距离最近的两个簇,直到所有点都在一个簇中。
- 分裂式: 从所有点在一个簇开始,逐步分裂距离最远的簇,直到每个点都成为一个簇。
sklearn 提供了 AgglomerativeClustering,它实现了凝聚式层次聚类。
代码实现
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import AgglomerativeClustering
from sklearn.datasets import make_blobs
from scipy.cluster.hierarchy import dendrogram, linkage
# 1. 生成示例数据
X, y_true = make_blobs(n_samples=100, centers=3, cluster_std=1.0, random_state=42)
# 2. 创建并训练层次聚类模型
# n_clusters: 簇的数量
# linkage: 簇间距离的计算方法 ('ward', 'complete', 'average', 'single')
# 'ward' 通常效果最好,它倾向于生成大小相似的簇
hc = AgglomerativeClustering(n_clusters=3, linkage='ward')
labels = hc.fit_predict(X)
# 3. 可视化聚类结果
plt.figure(figsize=(12, 5))
# 子图1: 聚类结果
plt.subplot(1, 2, 1)
plt.scatter(X[:, 0], X[:, 1], c=labels, s=50, cmap='viridis')'Agglomerative Clustering Result')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
# 子图2: 树状图
# 需要计算链接矩阵
plt.subplot(1, 2, 2)
linkage_matrix = linkage(X, method='ward')
dendrogram(linkage_matrix)'Dendrogram')
plt.xlabel('Sample index')
plt.ylabel('Distance')
plt.tight_layout()
plt.show()
树状图可以帮助你直观地看到簇是如何一步步合并的,从而帮助你决定最终的簇数量。
高斯混合模型
原理
GMM 假设数据是由多个高斯分布(正态分布)混合生成的,它不是为每个点分配一个硬标签,而是计算每个点属于每个高斯分布的概率,因此是一种软聚类方法,它使用 EM (Expectation-Maximization) 算法进行参数估计。
代码实现
import numpy as np
import matplotlib.pyplot as plt
from sklearn.mixture import GaussianMixture
from sklearn.datasets import make_blobs
# 1. 生成示例数据
X, y_true = make_blobs(n_samples=300, centers=3, cluster_std=1.2, random_state=42)
# 2. 创建并训练 GMM 模型
# n_components: 高斯分布(簇)的数量
# random_state: 随机种子
gmm = GaussianMixture(n_components=3, random_state=42)
gmm.fit(X)
# 3. 获取聚类结果
# predict() 返回概率最大的硬标签
labels = gmm.predict(X)
# predict_proba() 返回每个样本属于每个簇的概率
probs = gmm.predict_proba(X)
# 4. 可视化结果
plt.figure(figsize=(12, 5))
# 子图1: 聚类结果
plt.subplot(1, 2, 1)
plt.scatter(X[:, 0], X[:, 1], c=labels, s=50, cmap='viridis')'GMM Clustering Result')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
# 子图2: 概率可视化(用点的大小表示概率)
plt.subplot(1, 2, 2)
size = 50 * probs.max(axis=1) ** 2 # 取最大概率的平方来缩放点的大小
plt.scatter(X[:, 0], X[:, 1], c=labels, s=size, cmap='viridis', alpha=0.4)'Cluster Probabilities (Size = Confidence)')
plt.xlabel('Feature 1')
plt.ylabel('Feature 2')
plt.tight_layout()
plt.show()
聚类评估指标
因为没有真实标签,评估聚类质量比较困难,通常分为两类:
内部指标
评估聚类结果本身的质量,不依赖外部标签。
-
轮廓系数:
- 范围在
[-1, 1]之间。 - 值越大,说明样本同簇内相似度高,簇间相似度低,聚类效果越好。
sklearn.metrics.silhouette_score
- 范围在
-
Calinski-Harabasz 指数 (方差比准则):
- 值越大,说明簇间离散度越大,簇内离散度越小,聚类效果越好。
sklearn.metrics.calinski_harabasz_score
from sklearn.metrics import silhouette_score, calinski_harabasz_score
# 假设 X 是数据,labels 是聚类标签
silhouette_avg = silhouette_score(X, labels)
ch_score = calinski_harabasz_score(X, labels)
print(f"Silhouette Score: {silhouette_avg:.3f}")
print(f"Calinski-Harabasz Score: {ch_score:.3f}")
外部指标
当有真实标签时,可以评估聚类结果与真实标签的吻合程度。
-
调整兰德指数:
- 范围在
[-1, 1]之间,1 表示完美匹配。 sklearn.metrics.adjusted_rand_score
- 范围在
-
调整互信息:
- 范围在
[0, 1]之间,1 表示完美匹配。 sklearn.metrics.adjusted_mutual_info_score
- 范围在
from sklearn.metrics import adjusted_rand_score, adjusted_mutual_info_score
# y_true 是真实标签,labels 是聚类标签
ari = adjusted_rand_score(y_true, labels)
ami = adjusted_mutual_info_score(y_true, labels)
print(f"Adjusted Rand Index: {ari:.3f}")
print(f"Adjusted Mutual Info: {ami:.3f}")
聚类工作流总结
一个完整的聚类分析流程通常如下:
-
数据准备与探索:
- 加载数据。
- 数据清洗(处理缺失值、异常值)。
- 特征缩放:非常重要! 聚类算法(尤其是基于距离的)对特征的尺度非常敏感,务必使用
StandardScaler或MinMaxScaler对数据进行标准化或归一化。
-
选择算法和参数:
- 根据数据特点(形状、大小、是否有噪声)选择合适的算法。
- 使用肘部法则、轮廓系数等方法确定 K 值(对于 K-Means, GMM)。
- 调整算法参数(如 DBSCAN 的
eps和min_samples)。
-
训练模型:
- 使用
fit()方法训练模型。
- 使用
-
评估结果:
- 使用内部指标(轮廓系数等)评估聚类质量。
- 如果有真实标签,使用外部指标(ARI 等)进行评估。
- 可视化:降维(如 PCA, t-SNE)后绘制聚类结果,直观判断。
-
解释与应用:
- 分析每个簇的特征,为业务决策提供依据。
- 将聚类结果用于下游任务,如客户分群、图像分割等。
希望这份详细的指南能帮助你掌握 sklearn 中的聚类算法!